- 用PudMed搜尋"Clostridium difficile R20291 carbon storage regulator ",看有沒有相關的文獻、作者、趨勢分析,但由於此蛋白在 Clostridium difficile 中並沒有被廣泛研究過,因此搜尋結果只有一個關於Gene的item顯示。

CsrA gene的資料:網頁下拉可看到更多相關資料,按"GenBank"最下面有此基因的序列(213bp)。


也可看CsrA protein的資訊。

- 建立親緣關係樹將Clostridium difficile carbon storage regulator A利用NCBI BLAST "nucleotide blast"做Blast。
貼上Clostridium difficile 的(carbon storage regulator A) CsrA基因序列,並且在 " Program selection"選擇 More dissimilar sequences (discontiguous megablast)可搜尋到較多的序列,選擇"Show results in a new window"、"BLAST"。
幾分鐘後BLAST的結果出爐,系統會列出一個清單,上面顯示相似的基因序列以及其物種,依相似性由高到低排列。
由於資料並不多(只有12筆),因此只選其中較感興趣的7筆CsrA序列相似的菌株做比較,按Download, FASTA (aligned sequences),下載後用BioEdit程式開啟檔案。
(!!!序列名稱盡可能簡短,避免後續建立親緣關係樹時造成程式誤會)
點選Accessory Application > ClustalW Multiple Alignment > RunClustalW> 完成alignment 。
儲存檔案:File > Save as > phylip-3.69資料夾 > exe資料夾 > 檔名改Infile.phy (Phylip 4 格式)
依流程表做 Tree 打開 phylip-3.69中的 exe資料夾,將之前的 infile.phy的副檔名刪除。
打開 phylip-3.69中的 exe資料夾,將之前的 infile.phy的副檔名刪除。
開啟 seqboot.exe,會出現以下畫面,再依序輸入指令: R > Enter > Numbers of replicates > 250 > Y > Enter > Random number seed > 1
接著會得到一個outfile檔案,將原本infile檔名改成其他名稱(例:CsrA-01),再將outfile改成infile檔名,方便下個程式開啟。
打開 protdist.exe程式,如下圖所示,輸入指令: M > multiple data sets (type D) > How many data sets > 250 > Y
等待系統作業,之後會得到另一個outfile,將上個infile檔名改成其他名稱(CsrA-02),再將outfile改成infile檔名,方便下個程式開啟。
打開 neighbor.exe程式,輸入指令: M > How many data sets > 250 > Random number seed > 1 > Y,之後會得到outfile及outtree兩個檔案。
把之前infile及outfile檔名都改掉,並將 outtree檔名改成 intree,方便下個程式開啟。
開啟consense.exe程式,輸入指令"Y"後程式會產生 "outfile" 及 "outtree" 兩個檔案。
可用兩種程式開啟outtree,"TreeViewX"和"hypertree-1.2.2"。

- TMev分析圖首先要先取得欲探究的microarray資料,用 NCBI GEO DataSets 搜尋 E.coli CsrA ,並得到下圖。

接著把網頁下拉,選擇"MINiML formatted family file(s)",下載檔案然後再解壓縮,不過因為技術上的問題,檔案解壓縮後呈現各自的 txt檔,檔案因此無法合併匯入Mev中進行分析,嘗試轉換在excel中也無法開啟,在.txt檔中合併也沒辦法成功匯入Mev中,之後若有解決辦法,則會再更新方法,本次就先以老師上課的範例最為使用說明。
開啟microarray軟體MeV_4_8_1,打開老師給的範例data,步驟:File > Select file loader > Other fomat files > GenePix format files > Browse > 選擇資料夾 > Add all > load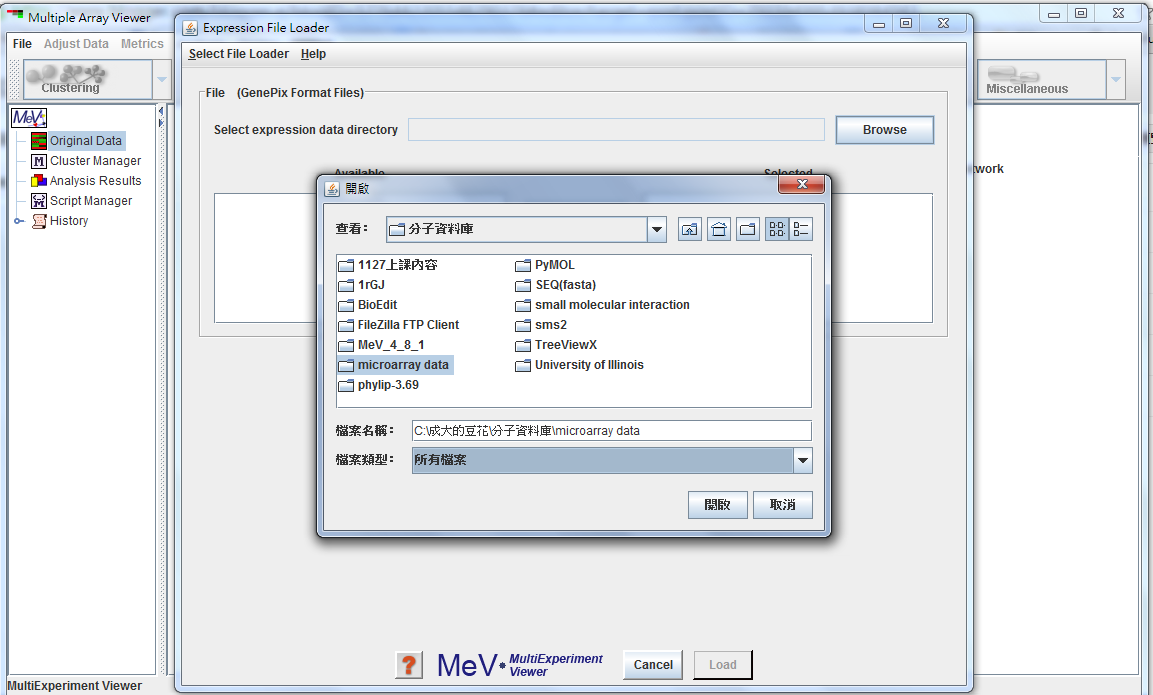

先 Normalization,讓基準點相同再做比較,就像是賽馬時先讓馬兒的體重相當。步驟:Adjust Data > Normalization > Total intensity。
比較data之間的相似程度,步驟:Clustering > Hierarchical Clustering > Pearson correlation,由此可找出有興趣的gene看相似度,但沒有分群。

若要有分群,步驟:Clustering > k-Means/ Medians Clustering,自己設定要分成幾群,此次就設定分10群。

也可以由電腦決定要分成幾群,用密度法的方式,離中心(mean)最近的去分組。步驟:Clustering > CAST (Cluster Affinity Search Technique),此例電腦分成17組Cluster。
查看哪些基因的表現之間最像,步驟:Analysis > Visualization > Gene Distance Matrix (GDM),圖示由黑到紅色漸層,越黑代表越相似。

可以按想看的gene,會跳出視窗顯示哪個gene與其最相似。例如:點下圖白色框的位置(B1, CTGF),再按"Expression graph"可以以折線圖方式表現。


KMC和CAST的比較,除了直接看Images外,還有"Centroid Graphs"、"Expression Graphs"、"Table views"的顯示方式。


- STRING (蛋白質交互作用預測)
進入首頁輸入蛋白質名稱或是序列,organism 的地方輸入目標物種。這裡以Clostridium difficile的CsrA為例。
出現 target protein和其他有相關的protein,點選後可查看這些蛋白的詳細資料。
圖的下方有一排功能可使用,例如:按"more"可顯示更多關聯性蛋白。
Views的地方可以查看各項詳細資料。
按下"Summary Network"圖片上方會出現一列功能表。
Enrichment > GO Biological Processes、GO Molecular Functions、GO Cellular Components,可分別去看不同性質的GO關係圖形。
例如:GO Molecular Functions > DNA binding > 紅色球所顯示。
BP
CC
MF
- CsrA蛋白結構圖
直接在搜尋的地方打上" Carbon Storage Regulator protein CsrA"。
右側的3D模型圖可點開,此處提到此蛋白的著色是隨著彩虹顏色的漸層表示N-terminal到 C-terminal。
也可以點選"3D View"可利用Java軟體看到更多面相 (拉遠、拉近、旋轉 )蛋白質結構。
開啟 PyMOL,點選 Plugin>PDB loader service,輸入"1Y00",即可叫出CsrA蛋白結構圖。

在 H (Hidden) 的地方點選"lines",S (Show) 選"Cartoon",呈現卡通的3D結構圖。
可在 Display選 background > white,讓背景呈現白色。
在右上方選擇"Ray"使結構看起來更鮮明、圓滑。
在 PyMOL >輸入"color red, chain A",可幫蛋白質著色,將蛋白質一不同的chain (A,B) 著上不同的顏色。
在Display選"sequence"可顯示蛋白質的胺基酸序列。
在PyMOL> 輸入"creat chain A, chain A",即可在物件中選擇不同的顯示方式。
例如:讓 chain A show > as "lines"。(其他還有sticks, ribbon, spheres)
在 1Y00 的Action(A)選擇 generate> vacuum electrostatic> protein contact potential (local),即可顯示蛋白區域的帶電性。
- miRNA binding site 預測
開啟 RNA22 v2 網站https://cm.jefferson.edu/rna22v2/ 選擇view pre-computed static RNA22 v2 predictions,再選HOMO SAPIENS>輸入Gene Name"NOD2" > Submit
選擇 View Predictions as cDNA map ,可以查看可能的miRNA位置及其序列。
已知miRNA後,RNAfold web sever 此網站 http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi可用來預測microRNA可能的structure。









沒有留言:
張貼留言